原副标题:中南大学自然生态大学刘建全科学研究项目组应邀该文解构和较为两套DNA水准的被子真菌形态学

被子真菌又名开花真菌,约有30余种,是亚种多元性程度最高、分布最广、适应性能力最强的真菌种属,主导了地球的陆地和水生自然生态系,为昆虫、两栖作战爬行类、鸟类和哺乳动物动物的多元性提供更多了重要的生境,也为人类提供更多了食物、能量等多种基本上物质条件(Lughadha et al. 2016; Kress et al. 2022)。从居维叶开始,科学家就为阐明被子真菌多元性科学研究中的核心环节––整座种属的形态学,即各种属间的亲缘亲密关系,开展了各种确凿证据的收集和各种类型的科学研究,但始终没有得到化解。而DNA统计数据被认为是复建被子真菌形态学最为关键性和最有希望的确凿证据和手段。被子真菌包括三种相同遗传模式的DNA: 核DNA、细胞器DNA和真核细胞DNA,全套DNA中的部份DNA或DNA短片的字符串突变都被用作形态学的创建 (Qiu et al. 1999)。近年来,也依次选用全套DNA的DNA水准的字符串突变构筑了被子真菌主要专业课程的形态学亲密关系;但是,两套DNA中部份或代表整座DNA水准的DNA字符串突变构筑的形态学亲密关系间仍然存在较大武装冲突。在这些科学研究中,各科、目所选用的指标性亚种相同,从而使科学研究统计数据和科学研究结论具有科学研究材料的不容较为性。特别是,核DNA水准的形态学大都是选用mRNA组或精简DNA抽取的相混字符串构筑,而不是倚赖 de novo DNA相混字符串的被子真菌形态学复建。随着质谱定序技术的快速发展,已有 de novo DNA的被子真菌亚种数量越来越多,为选用同样的指标性亚种,构筑两套DNA水准的形态学,创建或者说象征意义上的DNA被子真菌形态学 (Genome-scale angiosperm phylogeny, GAP) 提供更多了可能性。
JIPB近日在线刊登了 中南大学自然生态大学刘建全科学研究项目组专文“ Genome-scale angiosperm phylogenies based on nuclear, plastome, and mitochondrial datasets”的应邀该文 (https://doi.org/10.1111/jipb.13455)。该科学研究选用 de novo DNA和现代形态学裨益方法,解构和较为了两套DNA水准的DNA被子真菌形态学 (GAPs)。 该文汇整了近20年刊登的366个高效率被子真菌de novo DNA,包含241属、97科和43目。根据两套DNA类型的全DNA水准的字符串变化,第一次将同一亚种的两套DNA各自的相混字符串依次用作被子真菌形态学分析,构筑了或者说象征意义上的DNA被子真菌形态学(GAP)。与mRNA组或精简DNA统计数据相比 (如Baker et al. 2022), de novo DNA中抽取了更准确、更多的“相混”核DNA字符串;也避免了相同DNA较为时,选用相同亚种引致的不容较为性;还将科学研究结论与如前所述更大样本覆盖率、但只倚赖一种DNA统计数据的结论进行了较为 (Li et al. 2019; Baker et al. 2022)。科学研究结论阐明了相同DNA统计数据形态学武装冲突中的关键性结点和关键性种属(图1),并对其可能的原因,如不全然系谱甄选、繁育和DNA减半引致的非嫡系相混DNA抽取等进行了讨论。同时,也选用多化石残基,如前所述核DNA统计数据创建了被子真菌关键性结点的时间分化框架(图2)。该科学研究提供更多了第三版的DNA被子真菌形态学,为将来如前所述更多亚种 de novo DNA创建的GAP提供更多了基本上框架。最后,也指出未来要全然阐明被子真菌形态学亲密关系、化解现有相同DNA统计数据间的形态学武装冲突,应选用TNUMBERETDATE核DNA代替现代的嫡系相混单、低复本核DNA用作构筑形态学树,以及选用核DNA结构突变,即染色体无腺演化等新统计数据、新方法等来改变现状发现的相同DNA统计数据的形态学武装冲突问题 (Sun et al. 2022b; Wang et al. 2022)。
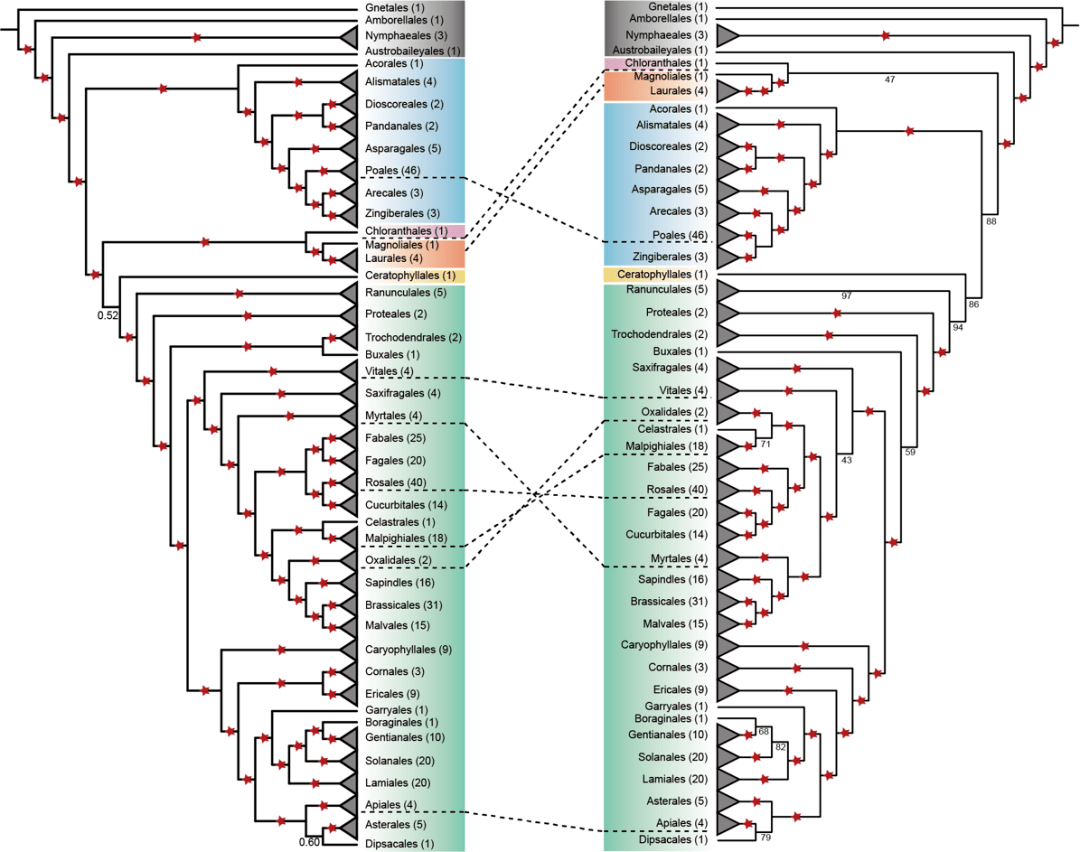
图1. 被子植物形态学框架和相同分类阶元存在的核质武装冲突。
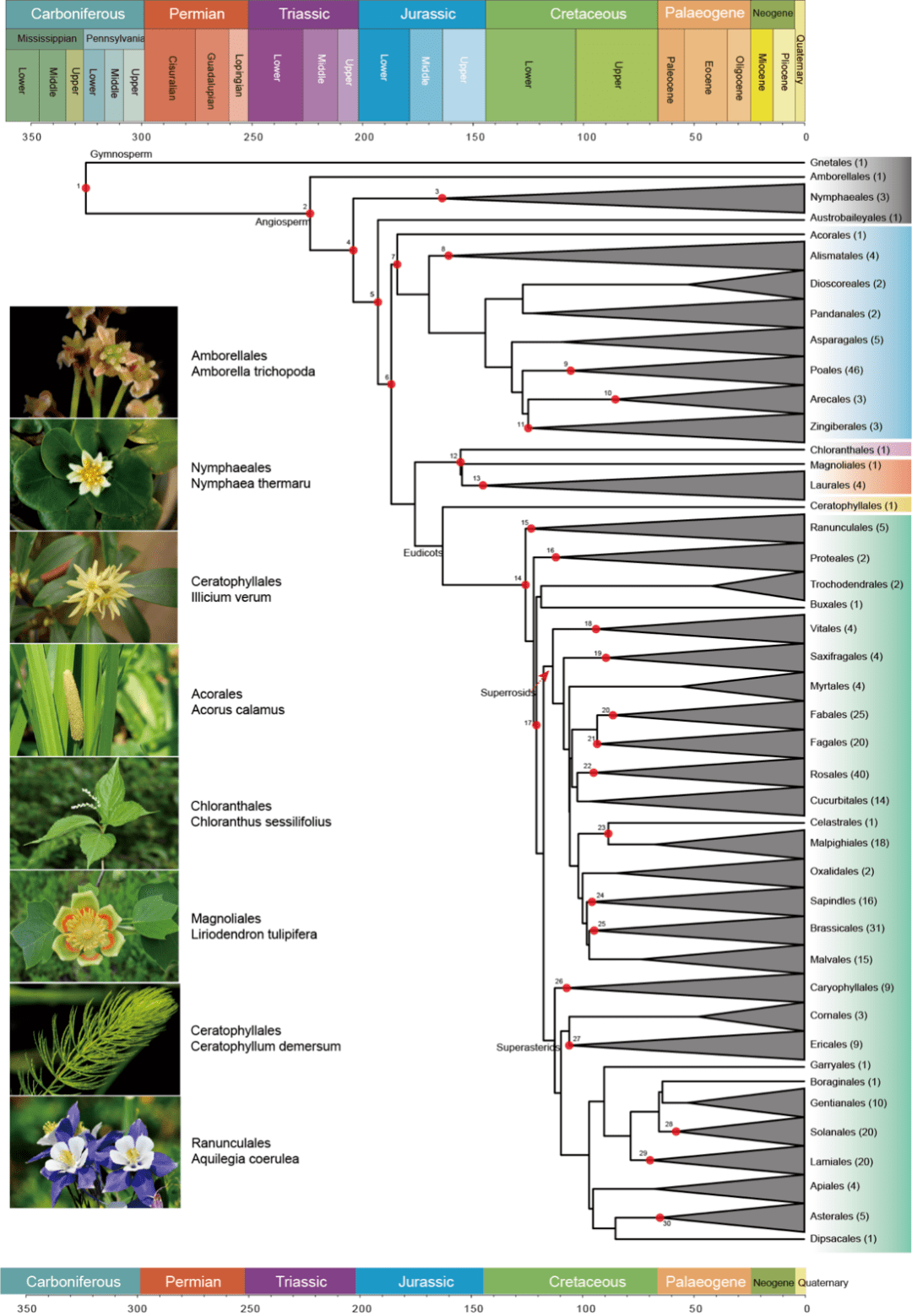
图2. 核DNA被子真菌主要分支的时间分化
刘建全科学研究项目组长期从事进化生物学和自然生态适应性科学研究。近年来利用DNA统计数据进行被子真菌早期演化历史的科学研究,刊登了一系列科学研究成果(Yang et al. 2020; Ma et al. 2021; Sun et al. 2022b);还针对选用TNUMBERETDATEDNA以及无腺演化进行形态学复建,开发了新分析软件WGDI(Sun et al. 2022a)。
参考文献:
Baker, W.J., Bailey, P., Barber, V., Barker, A., Bellot, S., Bishop, D., Botigue, L.R., Brewer, G., Carruthers, T., Clarkson, J.J., Cook, J., Cowan, R.S., Dodsworth, S., Epitawalage, N., Francoso, E., Gallego, B., Johnson, M.G., Kim, J.T., Leempoel, K., Maurin, O., Mcginnie, C., Pokorny, L., Roy, S., Stone, M., Toledo, E., Wickett, N.J., Zuntini, A.R., Eiserhardt, W.L., Kersey, P.J., Leitch, I.J., and Forest, F.(2022). A Comprehensive Phylogenomic Platform for Exploring the Angiosperm Tree of Life. Syst. Biol. 71: 301-319
Li, H.T., Yi, T.S., Gao, L.M., Ma, P.F., Zhang, T., Yang, J.B., Gitzendanner, M.A., Fritsch, P.W., Cai, J., Luo, Y., Wang, H., van der Bank, M., Zhang, S.D., Wang, Q.F., Wang, J., Zhang, Z.R., Fu, C.N., Yang, J., Hollingsworth, P.M., Chase, M.W., Soltis, D.E., Soltis, P.S., and Li, D.Z.(2019). Origin of angiosperms and the puzzle of the Jurassic gap. Nat. Plants 5: 461-470
Lughadha, E.N., Govaerts, R., Belyaeva, I., Black, N., Lindon, H., ALLKIN, R., MAGILL, R.E., and Nicolson, N.(2016). Counting counts: revised estimates of numbers of accepted species of flowering plants, seed plants, vascular plants and land plants with a review of other recent estimates. Phytotaxa 272: 82–88-82–88
Ma, J.X., Sun, P.C., Wang, D.D., Wang, Z.Y., Yang, J., Li, Y., Mu, W.J., Xu, R.P., Wu, Y., Dong, C.C., Shrestha, N., Liu, J.Q., and Yang, Y.Z.(2021). The Chloranthus sessilifolius genome provides insight into early diversification of angiosperms.Nat. Commun. 12:
Qiu, Y.L., Lee, J., Bernasconi-Quadroni, F., Soltis, D.E., Soltis, P.S., Zanis, M., Zimmer, E.A., Chen, Z., Savolainen, V., and Chase, M.W.(1999). The earliest angiosperms: evidence from mitochondrial, plastid and nuclear genomes. Nature 402: 404-407
Sun, P.C., Jiao, B.B., Yang, Y.Z., Shan, L.X., Li, T., Li, X.N., Xi, Z.X., Wang, X.Y., and Liu, J.Q.(2022a). WGDI: A user-friendly toolkit for evolutionary analyses of whole-genome duplications and ancestral karyotypes. Mol. Plant 15: 1841-1851
Sun, P.C., Yang, Y.Z., Liu, L., Xi, Z.X., Lin, H., Feng, L.D., Ma, J.X., Hu, H. Y., Bi, G.Q., Jiao, B.B., Wang, S., Yang, J., Mu, W.J., Zhu, M.J., Wang, Z.Y., Wang, X.Y., Charles, C.C.C., and Liu, J.Q. (2022b) Early diversification and karyotype evolution of flowering plants. Research Square Platform LLC,
Wang, Z., Li, Y., Sun, P., Zhu, M., Wang, D., Lu, Z., Hu, H., Xu, R., Zhang, J., Ma, J., Liu, J., and Yang, Y.(2022). A high-quality Buxus austro-yunnanensis (Buxales) genome provides new insights into karyotype evolution in early eudicots. BMC Biol. 20: 216
Yang, Y.Z., Sun, P.C., Lv, L.K., Wang, D.L., Ru, D.F., Li, Y., Ma, T., Zhang, L., Shen, X.X., Meng, F.B., Jiao, B.B., Shan, L.X., Liu, M., Wang, Q.F., Qin, Z.J., Xi, Z.X., Wang, X.Y., Davis, C.C., and Liu, J.Q.(2020). Prickly waterlily and rigid hornwort genomes shed light on early angiosperm evolution. Nat. Plants 6: 215-222
JIPB面向全球,刊发整合真菌生物学科学研究的重要创新成果,包括宏观和微观领域有创新性的重要科学研究论文、综述、突破性报道、新资源、新技术和评论性该文等。2021年2年SCI_IF: 9.106,位于真菌学TOP 4%,SCI的Q1区。Scopus统计数据库中CiteScore: 11.8,位于TOP 2.9%。JIPB位于中国科大学期刊分区生物学大类1区和真菌学小类1区,中国科协《真菌科学领域高效率期刊分级目录》T1级,并入选中国科技期刊卓越行动计划。 返回搜狐,查看更多
责任编辑:




还木有评论哦,快来抢沙发吧~